HumanPilot: primer estudio de transcriptómica a resolución espacial usando Visium
¿Prefieres ver un video en vez de leer? Checa este video explicativo 🎥
Hace unos años (en 2021) publicamos un estudio al cual nos referimos con un nombre muy genérico: HumanPilot (Maynard, Collado-Torres, Weber, Uytingco et al., 2021). En ese estudio revisado por pares, fuimos los primeros en usar una tecnología comercialmente disponible para generar datos transcriptómicos con resolución espacial. Se llama Visium y la vende 10x Genomics. ⚠️ Tomen en cuenta que️ empleados de esa compañía fueron coautores de nuestro estudio. Como trabajamos en el Institute Lieber para el Desarrollo del Cerebro, probamos Visium con muestras post mortem de cerebro humano. Expliqué la versión preimpresa (antes de la revisión por pares) de nuestro trabajo en este texto que escribí el 29 de febrero de 2020.
🔥off the press! 👀 our @biorxivpreprint on human 🧠brain @LieberInstitute spatial 🌌🔬transcriptomics data 🧬using Visium @10xGenomics🎉#spatialLIBD
— 🇲🇽 Leonardo Collado-Torres (@lcolladotor) February 29, 2020
🔍https://t.co/RTW0VscUKR
👩🏾💻https://t.co/bsg04XKONr
📚https://t.co/FJDOOzrAJ6
📦https://t.co/Au5jwADGhYhttps://t.co/PiWEDN9q2N pic.twitter.com/aWy0yLlR50
Ya fue suficiente de las palabras sobre especializadas. A continuación explicaré los puntos principales de porque nuestro estudio fue importante y de porque lo siguen citando varios años después.
¿Qué es una fruta? ¿Plátano? ¿Naranja?
Cuando estamos creciendo, no sabemos lo que es una naranja o un plátano. No sabemos lo que las hace frutas, y por qué son diferentes.
Si las ves, las tocas, las hueles, empiezas a notar diferencias entre ellas. En la ciencia, terminamos buscando formas sistemáticas y específicas de describir las cosas que nos rodean en la naturaleza. En este caso, dos frutas. Cuando observas una naranja y un plátano para explicar sus diferencias, puede que termines cortandolas en rebanadas.
Una vez que las rebanas, estás activamente acercándote y observando diferencias más finas. Puede ser que termines usando un microscopio para observar diferencias aún más pequeñas.
Midiendo niveles de actividad de genes
Ahora imagínate que quieres medir los niveles de actividad de genes 🧬. Algunas tecnologías previas pueden medir niveles de actividad de genes solo de bloques de tejido para llegar a obtener el material necesario. En la década de 2010 la tecnología para medir niveles de actividad de genes en células únicas o núcleos fue desarrollada, pero perdías el contexto espacial. Luego en 2020 la tecnología para medir niveles de actividad de genes reteniendo el contexto espacial fue nombrada como el método del año. El término específico es transcriptómica a resolución espacial.
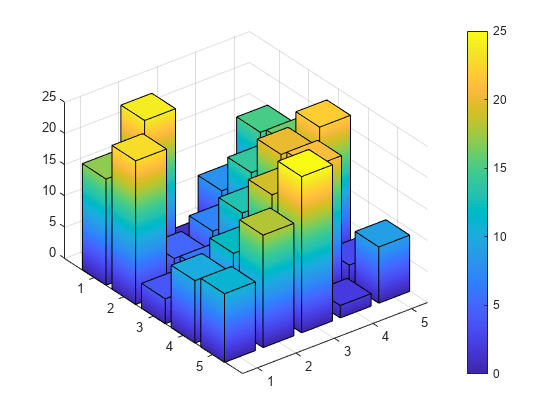
Específicamente, Visium funciona gracias a unos 5,000 círculos (llamados puntos, o “spots” en inglés) arreglados en forma de panal de miel de abeja 🐝, donde podemos medir los niveles de actividad de genes en cada uno de esos círculos. 10x Genomics tiene varios videos de cómo funciona su tecnología. Pero lo que aquí nos importa es que tenemos un mapa con coordenadas X y Y para el cual podemos hacer gráficas de barras en 3D como esta. Imagínate que esta es la gráfica para el gene 1 donde el mapa está plano y la altura de las barras nos indica que tan activo está el gene 1 en cada cuadrado.
Con Visium tenemos casi 5,000 puntos, o pares de (X, Y), y para cada uno de esos puntos podemos medir los niveles de actividad de unos 2,000 genes. Así que imagínate que tenemos 2,000 de estas gráficas de barras en 3D. Eso es un montón de datos y es emocionante pensar para qué podemos usar esta tecnología.
Cerebro humano
Tal y como estábamos hablando de plátanos y naranjas como dos tipos de frutas, pues, el cerebro tiene varias regiones. Cada una de estas regiones lleva a cabo funciones específicas, las regiones pueden llegar a ser bastante diferentes pero al fin y al cabo, son parte del cerebro. Tal y como los plátanos y las naranjas son muy diferentes pero siguen siendo frutas.
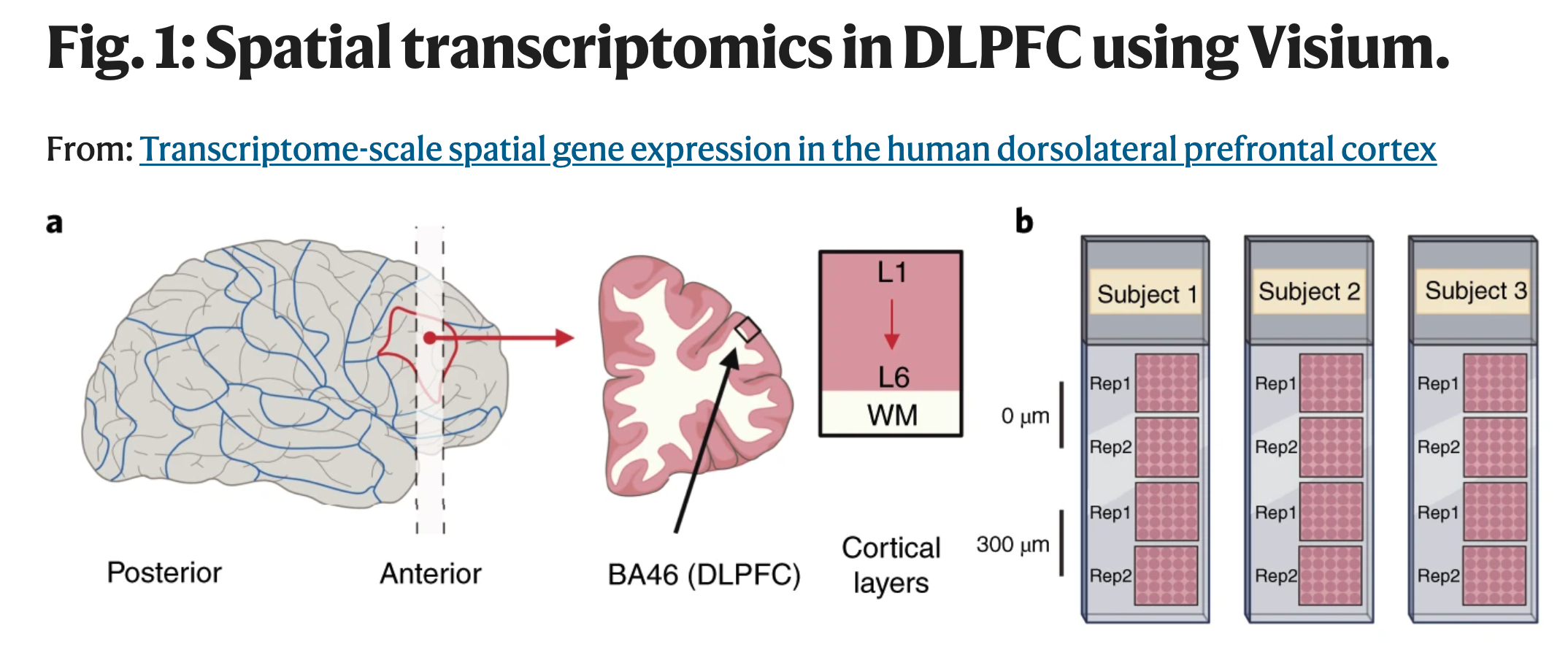
Ahora, los científicos son escépticos por entrenamiento. Así que con una nueva pero cara tecnología, lo primero que queríamos hacer era probarla con un pequeño estudio. Es decir, un estudio piloto. Nosotros trabajamos estudiando niveles de actividad de genes de muestras de cerebro humano, particularmente de tejido humano que fue donado a nuestro instituto con fines de investigación. Una región en la que estamos interesados se llama DLPFC (por sus siglas en inglés) y es importante porque hay diferencias entre individuos diagnosticados con esquizofrenia e individuos que no tienen ningún síntoma relacionado a la esquizofrenia (el grupo de controles neurotípicos) que han sigo ligadas a esta región del cerebro, entre otras razones.
Pero antes de que podamos distinguir a los dos grupos de donadores a nivel molecular, con el objetivo final de ayudar a las personas afectadas por desórdenes como la esquizofrenia, primero necesitamos entender las diferencias típicas entre controles neurotípicos. Tal y como podemos observar diferentes plátanos saludables y notar diferencias entre ellos.
Así fue que empezamos nuestro estudio HumanPilot. Generamos datos de tres controles neurotípicos ya que necesitas datos de más de un donante para tener alguna idea mínima de la variación biológica.
Pero porque Visium era una nueva tecnología en ese entonces, también generamos réplicas espacialmente adyacentes (dos pares por donante). Estas son réplicas técnicas en el sentido de que tomamos rebanadas contiguas de tejido. Piensa en dos pares de rebanas de pan que están a 10 micrómetros de distancia: generamos un par al inicio del pan y otro par 300 micrómetros después. Si, las cosas cambian aún a una distancia de 10 micrómetros, pero ojalá no cambian mucho, aunque si deberían de cambiar más 300 micrómetros después. Eso es lo que queríamos ver.
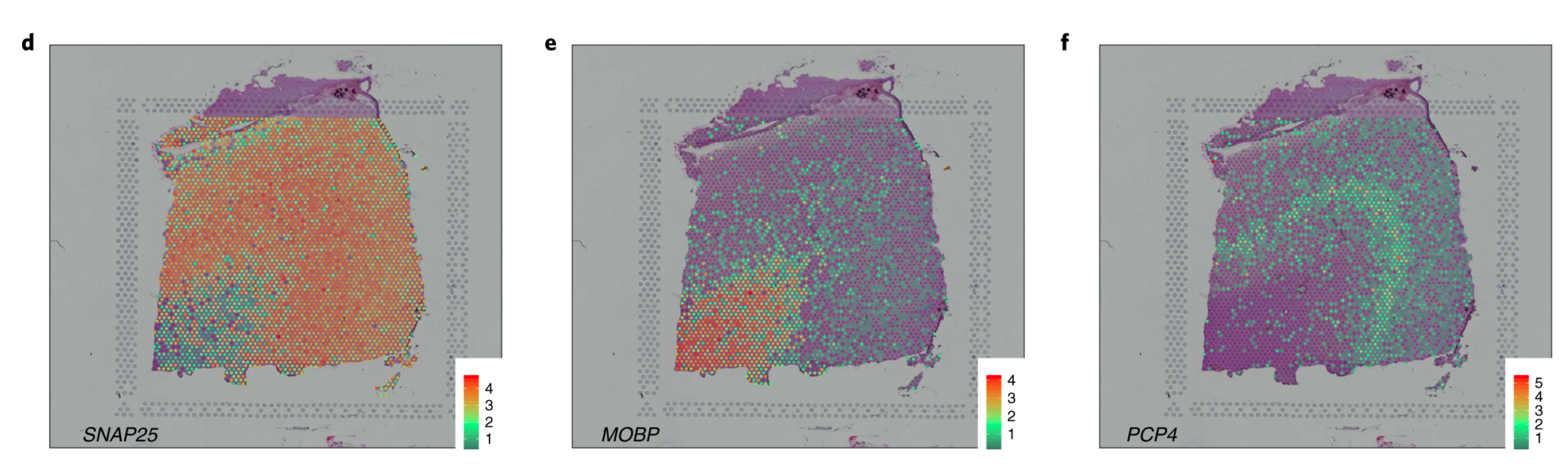
El DLPFC ha sido bastante bien estudiado y es muy conocido que el gene SNAP25 es más activo en las neuronas mientras que MOBP no está tan activo en neuronas. Cuando vimos los niveles de actividad de estos genes, pudimos ver que se complementaban muy bien tal y cómo el símbolo de Yin y Yang ☯. Así que estabamos muy emocionados.

También podíamos ver patrones más especializados en varios genes, tal como con el gene PCP4 que se usa para identificar la capa 5 del DLPFC. Algo importante que vimos es que este patrón era consistente entre los 6 pares de replicas especialmente adyacentes. Busca los patrones consistentes en la imagen.
¡Un montón de trabajo manual para reproducibir lo que ya se sabía!
Ahora que ya confiabamos en la tecnología, nos pudimos poner a trabajar. Empezamos visualizando patrones de niveles de actividad de genes para genes que previamente ya se habían identificado como marcadores para las capas de DLPFC. Algunos genes fueron identificados como marcadores usando otros organismos como el ratón 🐁. Algunos, por las propriedades de Visium, no los pudimos observar. Visium solo puede medir unos 2,000 genes por punto en vez de otras tecnologías que miden entre 20 y 30 mil genes pero que pierden el contexto espacial.
Our paper describing our package #spatialLIBD is finally out! 🎉🎉🎉
— Brenda Pardo (@PardoBree) April 30, 2021
spatialLIBD is an #rstats / @Bioconductor package to visualize spatial transcriptomics data.
⁰
This is especially exciting for me as it is my first paper as a first author 🦑.https://t.co/COW013x4GA
1/9 pic.twitter.com/xevIUg3IsA
Visualización de datos, particularmente con cualquier tecnología nueva, siempre es un reto. Así que desarrollamos una herramienta para visualizar estos datos y le dimos el nombre de spatialLIBD (Pardo, Spangler, Weber, Hicks et al., 2022). Construimos spatialLIBD de tal forma que personas de nuestro equipo pudieron asignar cada uno de los puntos de Visium de nuestras 12 rebanadas de tejido a una de 7 categorías: las capas de materia gris que van de la 1 a la 6, y la materia blanca. ¡Eso fue un montón de trabajo! Muchas otras personas ahora se refieren a esta anotación manual como la “verdad fundamental” pero sabemos que no lo es. En total tenemos 47,681 puntos, es decir un promedio de 3,973 puntos por rebanada de tejido. ¡Imagínate tener que etiquetar tantas frutas! Es probable que cometas algunos errores.
Eso fue una gran cantidad de trabajo, y es una de las razones fundamentales por las cuales nuestro estudio ha sido tan citado hasta ahora. Pero antes de que me adelante, ahora que ya tenemos todos los puntos asignados a su categoría correspondiente, podemos colapsar los datos y realizar una serie de análisis. Uno de ellos es usar análisis de componentes principales (PCA en inglés) para visualizar las diferentes capas y la materia blanca (WM en inglés) de nuestras 12 rebanadas de tejido. Para revisar la teoría matemática detrás de PCA, checa Wikipedia. Lo que aquí importa es que PC1 explica la mayoría de las diferencias, y que podemos ver a los puntos WM en negro del lado izquierdo claramente separados de los demás puntos. Luego en PC2 podemos ver a L1 en rosa hasta abajo del eje Y, seguido de los puntos azules de L2, los verdes de L3, los morados de L4, los amarillos de L5, y por último los naranjas de L6.
¡Eso fue asombroso! Si, la materia blanca es muy diferente de la materia gris (tal y cómo lo vemos en el PC1), pero también las capas de la materia gris son diferentes entre ellas de forma secuencial (como se nota en PC2). Todo esto ya se sabía antes de nuestro estudio, pero siempre uno se siente bien cuando puedes usar una nueva tecnología para reproducir resultados anteriores.
Encontrando y validando nuevos genes marcadores de capas de DLPFC
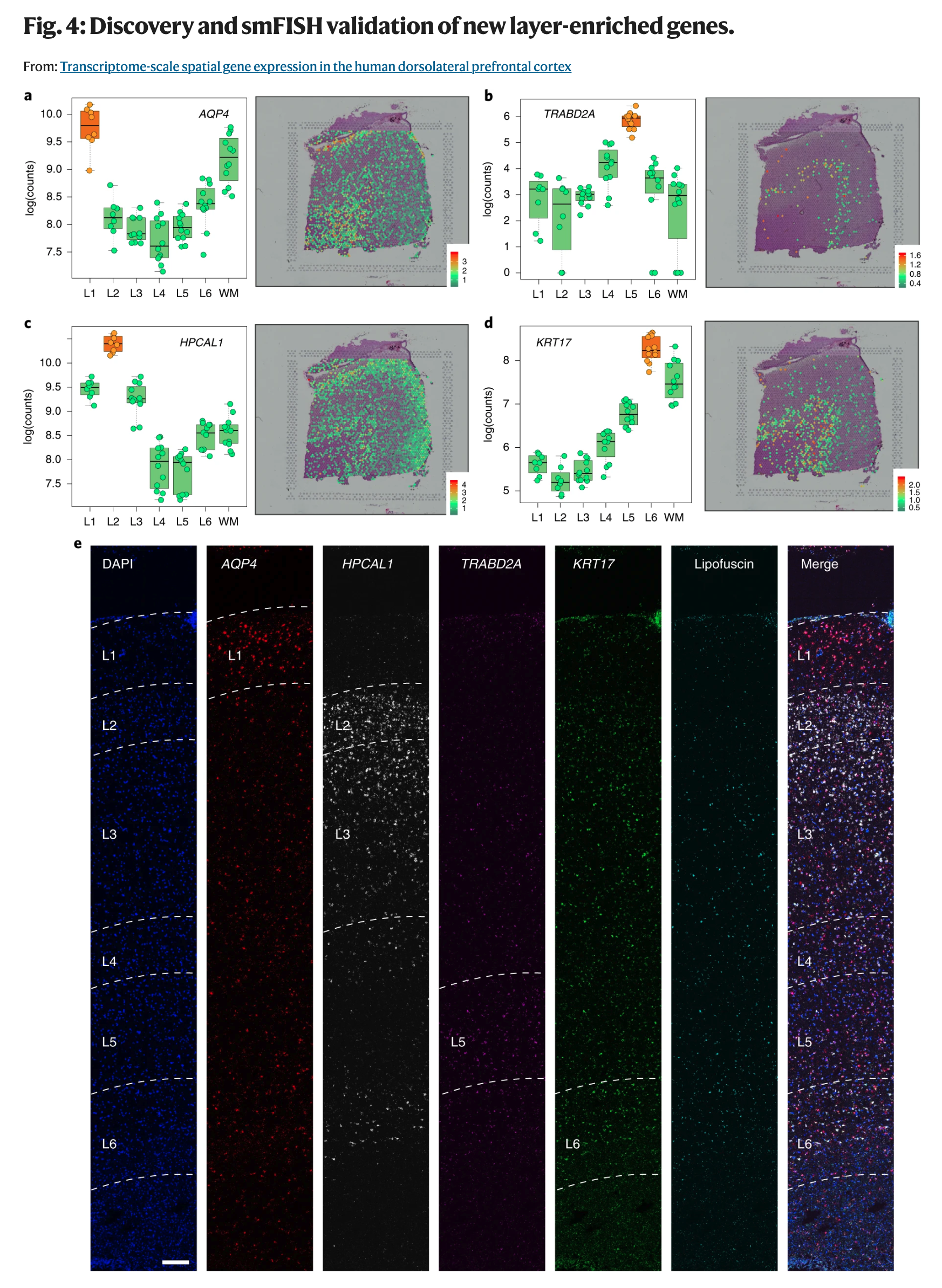
¿Eso fue todo? Claramente no. Otras tecnologías y experimentos laboriosos fueron necesarios para encontrar algunos genes marcadores de las capas de DLPFC. Pero ahora podemos identificar más, así que eso fue lo que hicimos. No solo pudimos encontrar nuevos genes que separan las capas entre ellas, pero pudimos validar nuestros resultados usando otros experimentos.
Esta imagen muestra como los nuevos genes marcadores tienen mayores niveles de actividad en las capas que marcan. Eso no significa que no estén activos en otros lados. Si te interesa encontrar genes marcadores que estén prendidos/apagados, hoy en día tenemos un método computacional para hacerlo que lo implementamos en nuestra herramienta de DeconvoBuddies.
We also propose and evaluate a new method for selecting cell type specific #markergenes for deconvolution: #MeanRatio implemented in #DeconvoBuddies (soon to be submited to @Bioconductor) #rstats
— Louise Huuki-Myers (@lahuuki) April 15, 2024
🛠️https://t.co/del9EjmBwQ pic.twitter.com/5PQk18yDrt
¿Y que nos dice su estudio HumanPilot sobre trastornos? ¿Nos ayuda a entenderlos?
Habiendo seguido nuestra historia hasta ahora, sabes que nuestros datos son de 3 donantes controles neurotípicos. No es como que podamos usar estos datos (n = 3) para encontrar las diferencias globales entre controles neurotípicos y personas que tienen algún trastorno cerebral. Pero lo que podemos hacer es preguntar, ¿hay un sobrelape significativo entre los genes marcadores de las capas de DLPFC y los genes que han sido previamente asociados a trastornos cerebrales?
Así que obtuvimos varias listas de genes de estudios previos con tamaños de muestra más grandes donde comparan controles neurotípicos contra individuos con ciertos trastornos cerebrales. Algunos de esos estudios están basados en niveles de actividad de genes, otros están basados en variantes de ADN que luego las ligan a genes basados en su proximidad. Algunos de los estudios que evaluamos fueron los que investigaron el trastorno de espectro autista (ASD en inglés). Lo que nos fascinó fue que dos estudios (SFARI y ASC102) mostraban un enriquecimiento similar entre genes asociados a ASD y los genes marcadores para las capas L2 y L5 que encontramos nosotros. Pero el estudio ASC102 se puede romper en dos sub-listas, la de ASD53 y la de DDID49, donde podimos ver que cada sub-lista estaba asociada solo con una de las capas (L5 y L2, respectivamente).
Así que transcriptómica a resolución espacial puede ser usada para localizar los elementos arquitectónicos de tejidos donde genes que han sido asociados a diversas enfermedades, trastornos, o grupos de interés biológico. Incluso otros estudios pilotos pequeños como el muestro podrían ayudarnos a entender como algunos de estos genes tiene patrones de actividad espacialmente distintos.
Ayudando el desarrollo de métodos, y aprovechandonos de este desarrollo
Sabíamos que no iba a ser el final. Nuestro HumanPilot era solo el inicio y para nuestros equipos de investigación, efectivamente empezó una ola 🌊 de nuevos estudios. Ahora estamos usando Visium y otras tecnologías para estudiar regiones de cerebro que no están tan bien estudiadas como el DLPFC, o para buscar diferencias entre grupos específicos de donantes. A la vez, esta no es nuestra primera vez que somos testigos al surgimiento de una nueva tecnología. Sabemos que cada tecnología tiene sus imperfecciones, sus sesgos, y sus fuentes de ruido de medición. Un reto en particular al cual queríamos ayudar era el proceso de anotar manualmente los puntos de Visium.
En nuestro set de datos, teníamos anotaciones manuales. Así que probamos varias formas de agrupar los puntos, conocido como clustering en inglés.
Cualquier método computacional de agrupación (clustering) va a generar resultados y gráficas bonitas. A ojo pelón, a veces uno puede detectar cuando un método generó malos resultados. Pero comparar a ojo los resultados de dos metodos decentes es muy complicado.
Usamos una medida conocida como índice de rand ajustado para hacer esta comparación, donde valores más altos son mejores. Hicimos una serie de cosas clave:
- Hicimos públicos los datos de nuestro estudio en cuando hicimos la versión preimpresa pública el 29 de febrero de 2020.
- Es decir, no nos esperamos a completar todo el proceso de revisión por pares (nuestro estudio finalmente fue publicado el 8 de febrero de 2021, casi un año después). Compartir de forma libre los datos de forma temprana ayuda a acelerar la ciencia.
- Hicimos facíl el proceso de accesar nuestros datos en un formato computacional fácil de usar.
- Específicamente en el formato
SingleCellExperimentde R/Bioconductor y que se pueden descargar con solo una función:spatialLIBD::fetch_data(). Hoy en día, proveemos los datos en el formatoSpatialExperimentque es más especializado.
- Específicamente en el formato
- Mostramos como comparar los resultados de agrupación (clustering) contra la anotación manual usando el índice de rand ajustado.
En unos meses, otras personas como los autores de BayesSpace el 5 de septiembre de 2020, usaron nuestros datos de HumanPilot para evaluar el rendimiento de sus sofisticados métodos de agrupación (clustering) que son sensibles al contexto espacial.
Ha sido especial ser testigos de esta rápida velocidad de desarollo. Hemos podido observar como otros usan nuestros datos para mejorar sus métodos, y ahora podemos salir beneficiados al usar esos nuevos métodos para nuevos set de datos de Visium que generamos sin tener que hacer el proceso laborioso de anotación manual.
Qué siguió
Naturalmente recibimos preguntas sobre que podría pasar si nuestro estudio piloto tuviera más datos. Sobre que pasa a lo largo del DLPFC (anterior, en medio, posterior). Hoy, nuestro siguiente estudio revisado por pares fue publicado con datos de 10 controles neurotípicos a lo largo del eje anterior-posterior para un total de 30 rebanadas de tejido con Visium. Próximamente explicaré este nuevo estudio, el cual llamamos spatialDLPFC. Mientras, Louise A. Huuki-Myers ya escribió una excelente descripción general sobre spatialDLPFC que les recomiendo que lean (solo disponible en inglés).
New in @sciencemagazine: our work from @LieberInstitute #spatialDLPFC applies #snRNAseq and #Visium spatial transcriptomic in the DLPFC to better understand anatomical structure and cellular populations in the human brain #PsychENCODE https://t.co/DKZqmG4YDi https://t.co/Tjp2OjTo63 pic.twitter.com/vQbjts2JtQ
— Louise Huuki-Myers (@lahuuki) May 23, 2024
Algo que quiero resaltar es que mi colega Kristen R. Maynard y yo fuimos co-primer autores de HumanPilot, y ahora somos autores co-correspondientes de spatialDLPFC. HumanPilot empezó en 2019 y fue publicado en 2021, mientras que empezamos spatialDLPFC en 2020 y salió publicado en 2024. Estos proyectos tiene ciclos largos y toman un pueblo entero, o una federación de pueblos, para ser completados y publicados. Así que quiero agradecer a todos los colegas pasados. ¡No podríamos haber llegado a donde estamos sin su trabajo! Gracias a todas las personas que han estado interesadas en nuestro trabajo y que lo siguen usando para aprender nuevas cosas. Nosotros también aprendemos a través de ustedes. ¡¡¡Gracias!!!
Self-assessment can be repetitive - or maybe not? 🤔 This caught my attention in recent spatial omics literature (but apparently not the authors'?)... "More of a comment than a question" that we need benchmarks. pic.twitter.com/KPRTf0vUxv
— Helena Lucia Crowell (@helucro) November 29, 2022
Ojalá ahora tengan una mejor idea sobre nuestro estudio HumanPilot y ahora puedan reconocer el origen de imágenes como las de arriba. Si quieren aprender más, pueden ver la siguiente presentación más detallada que hicieron Cynthia Soto y Daianna Gonzalez-Padilla. Son parte de mi equipo, pero no estuvieron involucradas en este estudio. Así que su presentación es de una perspectiva más fresca.
Finalmente, sientanse libres de revisar nuestro código y más detalles en https://github.com/LieberInstitute/HumanPilot.
¡Nos estamos viendo por ahí! 😊
Agradecimientos
Esta entrada en el blog fue posible gracias a:
Referencias
\[1\] C. Boettiger. knitcitations: Citations for ‘Knitr’ Markdown Files. R package version 1.0.12. 2021. URL: https://CRAN.R-project.org/package=knitcitations.
\[2\] K. R. Maynard, L. Collado-Torres, L. M. Weber, C. Uytingco, et al. “Transcriptome-scale spatial gene expression in the human dorsolateral prefrontal cortex”. In: Nature Neuroscience (2021). DOI: 10.1038/s41593-020-00787-0. URL: https://www.nature.com/articles/s41593-020-00787-0.
\[3\] A. Oleś. BiocStyle: Standard styles for vignettes and other Bioconductor documents. R package version 2.32.0. 2024. URL: https://github.com/Bioconductor/BiocStyle.
\[4\] B. Pardo, A. Spangler, L. M. Weber, S. C. Hicks, et al. “spatialLIBD: an R/Bioconductor package to visualize spatially-resolved transcriptomics data”. In: BMC Genomics (2022). DOI: 10.1186/s12864-022-08601-w. URL: https://doi.org/10.1186/s12864-022-08601-w.
\[5\] Y. Xie, A. P. Hill, and A. Thomas. blogdown: Creating Websites with R Markdown. Boca Raton, Florida: Chapman and Hall/CRC, 2017. ISBN: 978-0815363729. URL: https://bookdown.org/yihui/blogdown/.