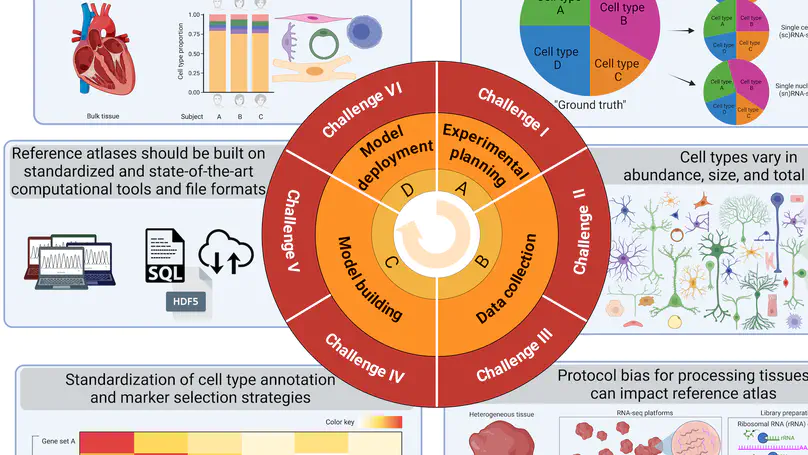
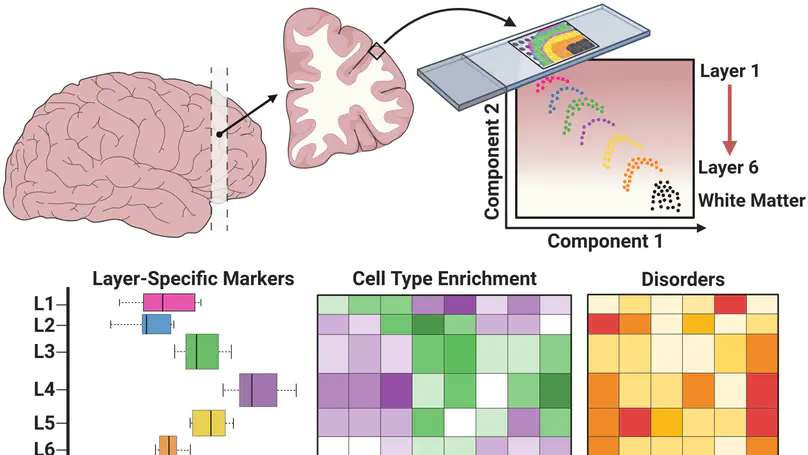
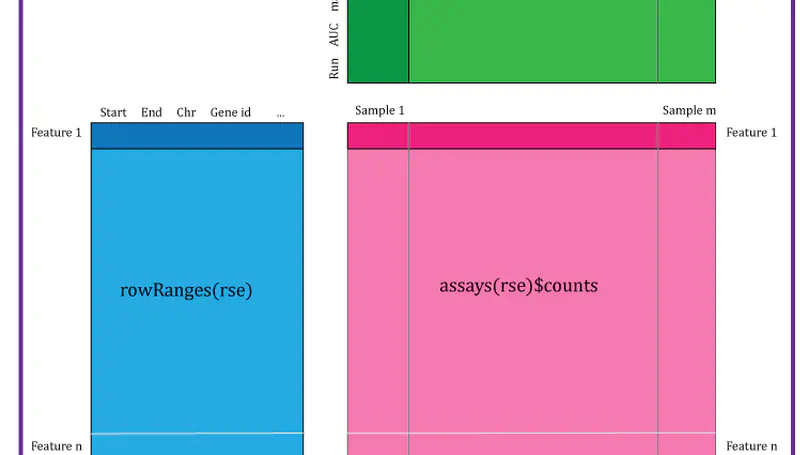
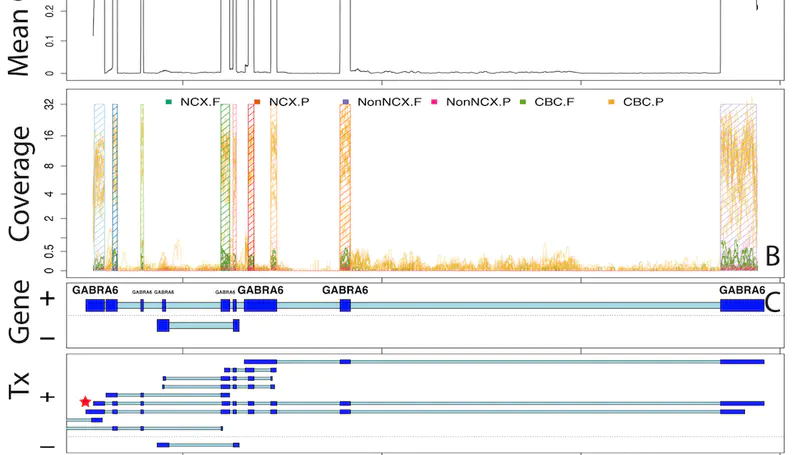
División de Neurociencia Traslacional, Ciencia de Datos I
JHPCE: lieber_lcolladotor
Instituto Lieber para el Desarrollo Cerebral
R/Bioconductor-powered Team Data Science

En el Instituto Lieber para el Desarrollo Cerebral (LIBD), como parte de la División de Neurociencia Traslacional, nuestro grupo trabaja para comprender las raíces y las firmas de las enfermedades (en particular, los trastornos psiquiátricos) al ampliar las dimensiones de la actividad genética. Logramos esto mediante el estudio de la expresión génica en todos los niveles de características de expresión (genes, exones, uniones exón-exón y regiones no anotadas) y mediante el uso de diferentes tecnologías de medición de la expresión génica (seq de ARN a granel, seq de ARN de células/núcleos individuales, y transcriptómica espacial) que proporcionan una mejor resolución biológica y localización de la expresión génica. Trabajamos en estrecha colaboración con colaboradores de LIBD, así como de la Universidad Johns Hopkins (JHU), la Universidad de Cambridge, y otras instituciones, lo que refleja el enfoque interdisciplinario y la diversidad de conocimientos necesarios para avanzar en nuestra comprensión de la biología de alto rendimiento.
Con el fin de proporcionar un entorno de investigación estimulante y de apoyo en LIBD, nuestro grupo ofrece sesiones de orientación sobre ciencia de datos abiertas a cualquier miembro del personal de LIBD. miembro y organizamos el LIBD rstats club, entre otras iniciativas. Nuestro sitio web del libro de documentación contiene más detalles sobre la incorporación al equipo, cómo solicitar ayuda, cursos internos, redacción de artículos científicos, autoría, archivos de configuración y mucho más.
Checa el contenido que compartimos
Constantemente creamos contenido nuevo para compartir lo que estamos aprendiendo o trabajando, lo que podría interesarle. En particular, nosotros:
- organizamos el LIBD rstats club: calendario del club
- discutimos artículos y software en nuestras reuniones de equipo: calendario del equipo
Únete al equipo
Si te interesa unirte al grupo R/Bioconductor-powered Team Data Science, consulta nuestras vacantes en el sitio web de oportunidades profesionales de LIBD. Es posible que te interese consultar nuestros resultados anónimos de la encuesta del equipo, que destaca algunas fortalezas pero también algunas debilidades y áreas que podemos mejorar.
Si no tenemos ningún puesto vacante, comunícate con Leonardo con tu CV, perfil de GitHub/GitLab/etc con software de código abierto y una breve descripción de por qué te interesa nuestro equipo.
Campos de interés
que nos emocionan
Proyectos
Presentaciones
de Leonardo
Esta sección solo está disponible en inglés.
Publicaciones recientes
y pósters de Leonardo
Esta sección solo está disponible en inglés.
Posts recientes de Leonardo en su blog
La mayoría de los posts de Leonardo están disponibles solo en inglés ya que le tomaría mucho tiempo traducirlos. Posts de la categoría rstats también son difundidos a través de RBloggers y R Weekly. También revisa el club de R de LIBD donde Leonardo contribuye posts.
Cursos impartidos por Leonardo
LIBD

2024
- Instructor del curso Introducción a análisis de datos de RNA-seq con Bioconductor para alumnos de la LCG-UNAM.
- Instructor invitado para un curso virtual organizado por EMBL-EBI.
- Instructor de una porción del curso Statistical Analysis of Genome Scale Data llevado a cabo en Cold Spring Harbor Laboratory.
- Instructor de la clase 140.776 Computación Estadística en la Johns Hopkins Bloomberg Escuela de Salud Pública.
2023
- Instructor del curso Introducción a análisis de datos de RNA-seq con Bioconductor para alumnos de la LCG-UNAM.
- Instructor de una porción del curso Statistical Analysis of Genome Scale Data llevado a cabo en Cold Spring Harbor Laboratory.
- Instructor de la clase 140.776 Computación Estadística en la Johns Hopkins Bloomberg Escuela de Salud Pública.
- Instructor invitado para CDSB 2023.
- Instructor invitado para este curso organizado con fondos de PROINNOVA para el Centro Universitario Los Altos.
2022
- Instructor del curso Introducción a análisis de datos de RNA-seq con Bioconductor para alumnos de la LCG-UNAM.
2021
- Organizador e instructor de CDSB 2021: Análisis de datos transcriptómicos de célula única (scRNA-seq) con R y Bioconductor.
- Instructor del curso Introducción a análisis de datos de RNA-seq con Bioconductor para alumnos de la LCG-UNAM.
- Instructor de la sesión de 2 horas sobre Análisis de scRNA-seq con Bioconductor, parte del taller Human Cell Atlas - Latin America.
- Instructor del mini curso Exploración interactiva de datos: plotly e iSEE para de la serie de mini cursos organizados por la CDSB, RMB y el NNB-CCG (UNAM).
2020
- Instructor para los talleres sobre R/Bioconductor y Ciencia de Datos en LIBD.
- Instructor y miembro del Comité Organizador del Taller CDSB 2020: Construyendo flujos de trabajo con RStudio y Bioconductor para datos transcriptómicos de célula única (scRNA-seq) (diapositivas en 🇲🇽)

- Instructor del curso Analyzing scRNA-seq data with Bioconductor para alumnos de la LCG-EJ-UNAM.
2019
- Instructor y miembro del Comité Organizador del Taller CDSB 2019: Cómo Crear y Ordenar Herramientas ‘Tidy’ (diapositivas en 🇲🇽)

2018
- Ponente principal y miembro del Comité Organizador del Taller Desarrolladores Latino Americanos de R/BioConductor 2018

2016
- Instructor de Stata y Bioestadística de un taller para personal docente de la Universidad de Kandahar, organizado por la Universidad Johns Hopkins.
- Instructor invitado para el curso Genomeeting 2016 course impartido en el INMEGEN, CDMX, México.
JHBSPH

2015-2016
- Asistente de enseñanza e instructor invitado para el curso Introducción a R para Investigadores de Salud Pública.
- Asistente de enseñanza para el curso de Métodos Estadísticos en Salud Pública I (140.621).
- Asistente líder de enseñanza para el curso de Métodos Estadísticos en Salud Pública II (140.622).
- Asistente de enseñanza para el proyecto MPH capstone dentro de la Maestría en Salud Pública.
2014-2015
- Asistente líder de enseñanza para los cursos de Métodos Estadísticos en Salud Pública I (140.621) y II (140.622).
- Asistente de enseñanza para el proyecto MPH capstone dentro de la Maestría en Salud Pública.
2013-2014
- Asistente de enseñanza para los cursos de Métodos Estadísticos en Salud Pública I (140.621) y II (140.622).
- Asistente de enseñanza para el proyecto MPH capstone dentro de la Maestría en Salud Pública. Desarrollé una interfaz con shiny para que los alumn@s se registraran para sesiones individuales de ayuda (código) y escribí un reporte sobre el número de sesiones de ayuda disponibles.
2012-2013
- Asistente de enseñanza para los cursos de Métodos Estadísticos en Salud Pública I (140.621), II (140.622), III (140.623), y IV (140.624).
UNAM

PDCB
Mientras trabajé en Winter Genomics enseñé dos cursos para alumnos del Programa de Doctorado de Ciencia Biomédicas (PDCB) de la UNAM.
- Analysis of High-Throughput Sequencing data with Bioconductor Aug-Dec 2010.
- Introduction to R and Biostatistics (along with two other teachers).
IBT
Mientras estuve dentro del Instituto de Biotecnología de la UNAM trabajando con Winter Genomics organicé dos cursos. Uno fue una serie de varios mini-cursos sobre bioinformática y biología, y el otro involucró a instructores de varias instituciones académicas.
- Introducción a R para biólog@s Oct-Nov 2009. Este mini-curso tiene bastante material para aprender a hacer gráficas con R. (diapositivas en 🇲🇽)
- Métodos Estadísticos y Analíticos de Datos Genómicos Enero 2010. Este curso de una semana fue compuesto por presentaciones introductorias sobre Perl, como usar un cluster, tecnologías de secuenciación masivas y otras relacionadas, R y Bioconductor, C, y biología. (diapositivas en 🇲🇽)
LCG
Impartí tres cursos durante mis años en la Licenciatura de Ciencias Genómicas (LCG). Cada uno de los cursos tiene su propio sitio web para organizar el material. Estos son:
- R/Bioconductor: curso intensivo Oct-Nov 2008 (diapositivas en 🇲🇽)
- Principios de Estadística Feb-Junio 2009 (diapositivas en 🇲🇽)
- Seminario III: R/Bioconductor Aug-Dec 2009 (diapositivas en 🇲🇽)
Curriculum vitae de Leonardo
Descarga el cv de Leonardo o explóralo a través de GitHub (solo está disponible en inglés).
Contacto
Si tienes preguntas sobre los paquetes de R/Bioconductor que mantiene Leonardo, por favor lée este post (en inglés). Si le envías un correo, simplemente te mandará la liga al mismo post.
- lcolladotor@gmail.com
- +1-301-450-2083
- 855 N. Wolfe, Oficina 382, Baltimore, MD 21205
- Entra al edificio Rangos, regístrate, sube el elevador al tercer piso, y vuele a registrarte en LIBD.
- Solicitar una cita
- Mándame un MD a Leonardo